Präsentation: Sechs Dinge über R
2025-09-08
1 Ein kurze Einführung in unser Arbeitsumgebung
1.1 RStudio, posit.cloud oder Positron?
R
Die Skript-Sprache mit der wir hier arbeiten werden. Dazu ein eigener Abschnitt später.RStudio (Desktop)
Eine Integrierte Entwicklungsumgeben (IDE) für R, die uns den Umgang sehr stark erleichtert.posit.cloud
Ursprunglich RStudio cloud, jetzt aber posit.cloud ist eine Cloud-Version von RStudio Desktop. Wir nutzen hier diesen Zugang, da er einen schnellern Zungang ermöglicht.Die Zukunft: positron
Der Nachfolger von RStudio Desktop ist in der Pipeline. Er heißt Positron
1.2 posit.cloud
Wir nutzen posit.cloud als Cloud-Version von R bzw. RStudio.
Für diese Einführung reicht das vollkommen aus. Sie sollten aber, wenn Sie öfters mit R arbeiten, darüber nachdenken R und R Studio oder Positron lokal auf ihren Rechner zu installieren. Anleitungen finden Sie dazu reichlich im Internet. Auch auf den auf den Seiten der FOM unter https://fomshinyapps.shinyapps.io/Installation-R-RStudio-mosaic/ bzw. https://raw.githubusercontent.com/luebby/Datenanalyse-mit-R/master/Installation/Install.pdf
1.3 Wo ist was?
Live: Die ersten Schritte in der posit.cloud!
2 Die Sprache R
2.1 Eine kurze Geschichte von R
Die Geschichte von R als Programmiersprache begann 1992 an der Universität Auckland in Neuseeland.
R wurde von den Statistikern Ross Ihaka und Robert Gentleman entwickelt.
Sie orientierten sich dabei stark an der Programmiersprache S, die in den Bell Laboratories in den 1970er Jahren entstanden war und für statistische Datenverarbeitung genutzt wurde.
R kann als freie Implementierung von S angesehen werden, wobei viele Programme für S auch mit R kompatibel sind.
Der Name „R“ leitet sich von den Anfangsbuchstaben der Vornamen der Entwickler Ross und Robert ab, gleichzeitig als Anspielung auf die Sprache S.
2.2 Eine kurze Geschichte von R
Die Motivation für die Entwicklung von R lag in der Verbesserung bestimmter Eigenschaften von S, wie etwa der Speicherverwaltung und der Flexibilität bei der Implementierung neuer analytischer Verfahren.
Für den Anfang wurde der Interpreter auf Basis von Scheme entwickelt, geschrieben wurde R in C, Fortran und der eigenen Sprache R.
Die Sprache wurde erstmals 1993 öffentlich zugänglich gemacht und seit 1995 unter der GNU General Public License als freie Software verbreitet.
Ein wichtiges Ereignis war 1997 die Gründung des R Development Core Teams sowie die Einrichtung des Comprehensive R Archive Network (CRAN), einer zentralen Plattform für Pakete und Erweiterungen.
R hat sich seitdem zu einer Standardprogrammiersprache für statistische Analysen entwickelt und wird weltweit in Wissenschaft und Wirtschaft eingesetzt.
Die Sprache bietet eine breite Palette an statistischen Methoden und Möglichkeiten zur grafischen Visualisierung und wird von einer großen Community stetig weiterentwickelt.
2.3 Noch ein paar Meilensteine in der Entwickliung
Bis 2010 prägten mehrere Meilensteine die Entwicklung der Programmiersprache R:
2000: Veröffentlichung von R Version 1.0, der ersten stabilen Version von R, die wichtige Funktionen wie Fehlerbehandlung (
try()), Datenrahmenbearbeitung (by(),merge()) enthielt.2001: Gründung von Bioconductor, ein Open-Source-Projekt für Genomdatenanalysen, das neben CRAN über 2000 weitere Pakete bereitstellt.
2007: Veröffentlichung von ggplot2, einem Paket basierend auf der Grammar of Graphics von Leland Wilkinson, entwickelt von Hadley Wickham. Dieses Paket revolutionierte die grafische Darstellung in R und wurde zu einem zentralen Bestandteil der R-Community und später des tidyverse.
2014: Das Paket dplyr mit seinen modernen Datenmanipulationsfunktionen wurde veröffentlicht. Eines der einflussreichsten Pakete im R-Ökosystem.
Diese Meilensteine zeigen eine kontinuierliche Verbesserung von R bezüglich Stabilität, Erweiterbarkeit und Benutzerfreundlichkeit.
2.4 Unser erstes R, ein R-Skript
Wir beginnen in dem wir uns in posit.cloud ein R-Skript Dokument erstellen und gemeinsam einige Schritte durch gehen um unser erstes (gemeinsames) R zu programmieren.
Wir beginnen mit den folgenden Zeilen:
Ausgabe:
Beachten Sie: Zuweisungen an eine Variable erfolgen durch “<-” oder “->”! An der Pfeilspitze steht immer die Variabel, auf der anderen Seite das, was sie Zuweisen wollen!
2.5 Wir machen etwas einfache Statistik
Zunächst bauen wir von Hand ein paar Daten auf, dazu erzeugen wir zwei Vektoren mit dem combine-Befehl c():
Jetzt können wir den BMI (also den Body Mass Index) einfach berechnen:
2.6 Noch ein paar Zeilen
Den Mittelwert können wir, quasi von Hand, berechnen in dem wir die Summe durch die Anzahl teilen:
Es gibt aber auch den Befehl mean() dafür:
2.7 Noch ein paar weitere Zeilen
Die Standardabweichung ist die Wurzel der Varianz, welche wiederum die durchschnittliche Summe der quadratischen Abweichungen vom Mittelwert ist:
Aber auch hierfür gibt es einfachere Befehle:
Oder noch kürzer mit dem Befehl sd() ( sd = standard derivation, zu deutsch Standardabweichung ):
2.8 Unsere erste Tabelle
Normalerweise werden in R Datenrahmen (data.frame) zur Speicherung von Tabellen benutzt.
Wir nutzen hier aber tibbles aus dem Paket tibble. Es sind im Prinzip Datenrahmen nur etwas moderner implementiert:
Bitte beachten Sie: Parameterzuweisungen, also Zuweisungen innerhalb von Funktionsaufrufen, erfolgen mit “=”!
2.9 Die Struktur einer Tabelle
Um die Struktur einer Tabelle darstellen zu können …
nutzen wir den Befehl str():
Oder moderner den Befehl glimpse():
2.10 Unterschied zum Standard-Datenrahmen
R hat auch eine Standard Implementierung für Datenrahmen. Der data.fram ist dem tibble sehr ähnlich.
Für Anfänger:in sind die unterschiedlichen Datentypen (tibble, data.frame, data.table, tbl, …) (annähernd) gleich zu benutzen. Auch wenn die Implementierungen sich teilweise starkt unterscheiden und es kontekt dann vor- bzw. nachteile geben kann.
2.11 Umwandeln von tibble nach data.frame und zurück
Wir können zwischen den beiden Datentypen umwandeln:
2.12 Unsere erste Graphik
Für Grafiken wollen wir ggplot() aus dem Paket ggplot2 benutzen. Dafür müssen wir zunächst das Paket laden:
Nun wollen wir ein Säulendiagramm erstellen:
2.13 Die Kennzahlen
Die Kennzahlen erhalten wir z.B. wie folgt:
2.14 Aufgaben
Erzeugen Sie eine neue Tabelle mit dem Namen tib2, welche die Spalten Groesse, Gewicht und BMI enthält.
Geben Sie die Mittelwerte aller drei Spalten aus.
Geben Sie ein Säulendiagramm des BMI an.
2.15 Das Paket dplyr
dplyr ist eine Grammatik zur Datenmanipulation und bietet eine konsistente Sammlung von Verben, die helfen, die häufigsten Herausforderungen bei der Datenbearbeitung zu lösen.
Ein Überblick:
mutate()fügt neue Variablen hinzu, die Funktionen bestehender Variablen sind.select()wählt Variablen basierend auf ihren Namen aus.filter()wählt Fälle basierend auf ihren Werten aus.summarise()reduziert mehrere Werte auf eine einzelne Zusammenfassung.arrange()verändert die Reihenfolge der Zeilen.
Diese Funktionen lassen sich mit group_by() kombinieren, wodurch jede Operation „nach Gruppe“ ausgeführt werden kann.
2.16 Backends zu dplyr
Neben der Arbeit mit Data Frames bzw. Tibbles ermöglicht dplyr auch einen effizienten und benutzerfreundlichen Umgang mit verschiedenen Backends. Nachfolgend dine Übersicht alternativer Backends:
arrow: Für Datensätze, die größer sind als der verfügbare Arbeitsspeicher – einschließlich solcher, die in entfernten Cloud-Speichern wie AWS S3 abgelegt sind.
dtplyr**: Für große Datensätze im Arbeitsspeicher. Übersetzt
dplyr-Code in leistungsstarkendata.table-Code.dbplyr: Für Daten, die in relationalen Datenbanken gespeichert sind. Wandelt
dplyr-Anweisungen in SQL-Abfragen um.duckplyr: Für die Nutzung von duckdb bei großen In-Memory-Datensätzen ohne zusätzliche Kopien. Übersetzt
dplyr-Code in effiziente duckdb-Abfragen und greift bei nicht möglicher Übersetzung automatisch auf R zurück.duckdb: Für große Datensätze, die dennoch vollständig auf einem lokalen Rechner verarbeitet werden können.
sparklyr: Für sehr große Datensätze, die in Apache Spark gespeichert sind.
2.17 Spalten zu Tabellen hinzufügen mit mutate()
Wir nutzen dazu das Paket dplyr und den Befehl mutate():
2.18 Ein Boxplot in zwei Dimensionen
Um die Unterschiede zwischen den weiblichen und männlichen Beobachtungen zu charaterisieren, können wir die Tabelle tib3 nutzen und Boxplots erstellen:
Horizontal:
Vertikal:
2.19 Aufgaben
Nutzen Sie die Tabelle tib3 und erstellen Sie einen Boxplot für die Varable Größe.
Nutzen Sie die Tabelle tib3 und erstellen Sie einen Boxplot für die Variable Größe, aber nach Geschlechtern getrennt!
2.20 Streudiagramme
Zur Darstellung von (vermuteten, linearen) Zusammenhängen zweier numerischer Variablen nutzen wir Streudiagramme (engl. Scatterplot, hier mit Hilfe des Geoms: geom_point()).
So können wir das Geom nutzen für die Darstellung:
Der Korrelationskoeffizient berechnet sich zu:
2.21 Eine lineare Regression
Eine lineare Regression scheint keine schlechte Idee zu sein.
Wir erstellen daher ein lineares Modell (lm()):
Eine ausführlichere Darstellung erhalten wir mit dem Befehls summary():
2.22 Eine Grafik statt nur die Kennzahlen
Wir können in das Streudiagramm auch die Regressionsgerade zeichnen lassen.
Ohne grossen Aufwand geht es so:
Mit etwas Aufwand geht mehr:
2.23 Tabellen und das Dollarzeichen
Intern speichert R Tabellen spaltenweise und nicht, wie zum Beispiel die meisten relationalen Datenbanken, zeilenweise!
Das ermöglicht einen schnellen Zugriff auf einzelne Spalten:
Ein Vektor mit den Daten aus der Spalte Gewicht:
Tabelle nur den Einträgen aus der Spalte Gewicht:
2.24 Spalten und Zeilen in R
Aber Vorsicht! Reines R kann ab und zu sehr ähnlich aussehen, aber unterschiedliche Ergebnisse liefern:
Liefert die erste Spalte!
Liefert die erste Zeile!
2.25 Spalten und Zeilen auswählen mit select() und filter() aus dplyr
Einfacher ist es mit dem Paket dplyr zu arbeiten.
So können wir Spalten mit dem Befehl select() auswählen bzw. selektieren:
Die Tabellenspalten können wir in einen Vektor umwandeln:
Zeilen werden mit dem Befehl filter() ausgewählt bzw. filtriert:
2.26 Zusammenfassungen von Daten mit summerise()
Wir wollen eine Zusammenfassung (eng. summarise) erstellen. Wir wollen wissen wie viele Beobachtungen (n) die Grundlage für den Mittelwert des Gewichts (mean) bzw des Median der Größe (median) sind:
2.27 Gruppieren mit group_by()
Mit dem Befehl group_by() können wir kategoriale Variablen (also Spalten) nutzen um einzelne Fälle (also Kategorien) zu unterscheiden:
2.28 Ein Datenaustauschformal CSV
CSV steht für Comma-Separated Values und ist ein einfaches, textbasiertes Dateiformat, mit dem tabellarische Daten gespeichert werden können. Jede Zeile in einer CSV-Datei entspricht einer Datenzeile; die einzelnen Datenfelder einer Zeile werden in der Regel durch ein Komma getrennt. CSV-Dateien werden häufig für den Datenaustausch zwischen verschiedenen Programmen, Datenbanken und Tabellenkalkulationen (wie Microsoft Excel oder R) verwendet. Aufgrund der Einfachheit ist das Format weit verbreitet, aber nicht streng standardisiert. Häufig sind die erste Zeile die Spaltennamen (Header) und jede Zeile danach enthält die eigentlichen Daten. Ein Beispiel für eine typische CSV-Struktur:
Name,Alter,Beruf,Stellenwert
Anna,34,Lehrerin,1.0
Bert,29,Ingenieur,0.5
Komma ist das Standardtrennzeichen, jedoch werden manchmal auch Semikolon, Tabulator oder andere Zeichen verwendet, abhängig von den regionalen und programmbezogenen Einstellungen.
2.29 Deutsche Besonderheiten führen zu CSV2
CSV2 ist eine Variation des CSV-Formats, die vor allem im deutschsprachigen Raum verwendet wird. Hierbei werden als Feldtrennzeichen Semikolons (;) statt Kommas genutzt, da das Komma oft als Dezimaltrennzeichen (z.B. bei Zahlenwerten) verwendet wird.
Hierbei erwartet die Funktion, dass Felder durch Semikolons getrennt sind und Zahlen das Komma als Dezimaltrennzeichen verwenden. Beispielhafter Aufbau einer CSV2-Datei:
Name;Alter;Beruf;Stellenwert
Anna;34;Lehrerin;1.0
Bert;29;Ingenieur;0.5
Das CSV2-Format eignet sich damit besonders für Kontinentaleuropa und Anwendungen, die diese Formatierung benötigen.
2.30 Daten aus einer Datei einlesen (CSV-Version)
Zum Einlesen einer CSV-Datei nutzen wir aus dem Paket readr den Befehle read_csv():
R hat nun die Spalten wir folgt gelesen:
2.31 Daten aus einer Datei einlesen (CSV2-Version)
Zum Einlesen einer CSV2-Datei nutzen wir aus dem Paket readr den Befehle read_csv2():
R hat nun die Spalten wir folgt gelesen:
2.32 Vergleich der eingelesenen Tabellen
Wir erhalten damit die selben Tabellen. Schauen wir kurz hinein:
Rows: 244
Columns: 7
$ total_bill <dbl> 16.99, 10.34, 21.01, 23.68, 24.59, 25.29, 8.77, 26.88, 15.0…
$ tip <dbl> 1.01, 1.66, 3.50, 3.31, 3.61, 4.71, 2.00, 3.12, 1.96, 3.23,…
$ sex <chr> "Female", "Male", "Male", "Male", "Female", "Male", "Male",…
$ smoker <chr> "No", "No", "No", "No", "No", "No", "No", "No", "No", "No",…
$ day <chr> "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Su…
$ time <chr> "Dinner", "Dinner", "Dinner", "Dinner", "Dinner", "Dinner",…
$ size <dbl> 2, 3, 3, 2, 4, 4, 2, 4, 2, 2, 2, 4, 2, 4, 2, 2, 3, 3, 3, 3,…Rows: 244
Columns: 7
$ total_bill <dbl> 16.99, 10.34, 21.01, 23.68, 24.59, 25.29, 8.77, 26.88, 15.0…
$ tip <dbl> 1.01, 1.66, 3.50, 3.31, 3.61, 4.71, 2.00, 3.12, 1.96, 3.23,…
$ sex <chr> "Female", "Male", "Male", "Male", "Female", "Male", "Male",…
$ smoker <chr> "No", "No", "No", "No", "No", "No", "No", "No", "No", "No",…
$ day <chr> "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Su…
$ time <chr> "Dinner", "Dinner", "Dinner", "Dinner", "Dinner", "Dinner",…
$ size <dbl> 2, 3, 3, 2, 4, 4, 2, 4, 2, 2, 2, 4, 2, 4, 2, 2, 3, 3, 3, 3,…2.33 Daten aus einer Datei einlesen (Excel-Version)
Um Daten aus einer Excel-Datei zu lesen brauchen wir das Paket readxl.
Wir können u.a. das Sheet “tips” auswählen, wenn das Dokument mehrere Tabellen enthält:
Wir haben nun die selben Daten in der Tabelle:
Rows: 244
Columns: 7
$ total_bill <dbl> 16.99, 10.34, 21.01, 23.68, 24.59, 25.29, 8.77, 26.88, 15.0…
$ tip <dbl> 1.01, 1.66, 3.50, 3.31, 3.61, 4.71, 2.00, 3.12, 1.96, 3.23,…
$ sex <chr> "Female", "Male", "Male", "Male", "Female", "Male", "Male",…
$ smoker <chr> "No", "No", "No", "No", "No", "No", "No", "No", "No", "No",…
$ day <chr> "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Sun", "Su…
$ time <chr> "Dinner", "Dinner", "Dinner", "Dinner", "Dinner", "Dinner",…
$ size <dbl> 2, 3, 3, 2, 4, 4, 2, 4, 2, 2, 2, 4, 2, 4, 2, 2, 3, 3, 3, 3,…2.34 Aufgaben
Versuchen Sie aus der Tabelle tips_csv die Anzahl der Frauen die Zahlen.
Tipp:sex == "Female") und deren Rechnungsbeträge (Tipp:total_bill) auszugebenGruppieren Sie nun die Ausgabe aus Aufgabe 1. so, dass sie nach Tagen ausgegeben wird.
Tipp:daysNutzen Sie die Hilfe um die Ausgabe der Größe der Rechnungsbeträge nach zu sortieren!
Einmal in aufsteigender Reihenfolge!
Einmal in absteigender Reihenfolge!
Tipp: Ist etwas schwerer! Suchen Sie mit der Hilfe im Paket
dplyrnach dem Befehlarrange().
2.35 Etwas den Speicher aufräumen!
Ab und zu ist einfach mal Zeit zum Aufräumen! – Und einer kleinen Pause!
2.36 Logische Vergleiche
Wir können in R zwei Zahlen miteinander vergleichen und feststellen, ob diese gleich oder ungleich sind. Wir können auch überprüfen, ob eine Zahl größer oder kleiner ist als eine andere Zahl. Eine solche Frage wird dann in R mit entweder Ja (TRUE) oder Nein (FALSE) beantwortet. Solche Vergleiche werden logische Vergleich genannt und sind auch nicht nur auf Zahlen beschränkt.
Wenn wir uns zum Beispiel dafür interessieren, ob die Zahl 5 größer ist als die Zahl 3 können wir dies mit dem Code 5 > 3 herausfinden:
Da 5 tatsächlich größer ist als 3, erhalten wir von R die Antwort TRUE.
2.37 Vergleichsoperatoren
Überblick:
| Symbol auf der Tastatur | Rechnung oder Operation |
|---|---|
== |
Gleich |
!= |
Ungleich |
> bzw. < |
Größer bzw. Kleiner |
>= bzw. <= |
Größergleich bzw. Kleinergleich |
Beispiele:
2.38 Die logische Verknüpfungen UND bzw. ODER
Neben den einfachen Vergleichsoperatoren ist es auch möglich, mehrere logische Vergleiche miteinander zu kombinieren. Als Verknüpfung sind vor allem das logische UND sowie das logische ODER relevant.
| Symbol auf der Tastatur | Operation | Bedeutung |
|---|---|---|
& |
logisches UND | Sind beide Vergleiche wahr? |
| |
logisches ODER | Ist mindestens eine der Vergleiche wahr? |
Als Antwort eines kombinierten logischen Vergleichs erhält man erneut entweder den Rückgabewert TRUE oder FALSE.
Tipp: Um sicherzugehen, dass R alle Symbole in der von uns gewünschten Reihenfolge auswertet, kann es sinnvoll sein Klammern zu setzen!
2.39 Beispiele
Beispiel 1:
Ist die Zahl 6 größer als 5 UND ist die Zahl 7 kleiner als 6?
Beispiel 2:
Ist die Zahl 7 größer als 5 ODER ist die Zahl 9 kleiner als 8 (oder beides)?
2.40 Datentypen
- numeric, integer, double
- Intern wird für Zahlen in R der Begriff numeric verwendet. Es gibt manchmal Situationen, in denen R weiter unterscheidet ob es sich um eine ganze Zahl (integer) oder eine Dezimalzahl (double) handelt. Mit jedem dieser drei Datentypen können mathematische Berechnungen vorgenommen werden. Für unsere Anwendungen ist es in der Regel egal, ob eine Zahl von R als numeric, integer oder double verstanden wird. Wir werden die Unterschiede daher nicht im Detail besprechen.
- logical
-
Wir haben gesehen, dassnach einem logischen Vergleich eine Information erhalten, die nur zwei Ausprägungen haben kann:
TRUEoderFALSE. Diese Informationen haben in R den Datentyp logical. Ist für einen Wert nun der Datentyp logical hinterlegt, weiß R, dass eben nur diese beiden Ausprägungen möglich sind.
2.41 Datentypen
- character (string)
- Eine weitere wichtige Funktion von R ist der Umgang mit Texten. Ein Text kann dabei unterschiedlich lang sein (ein Buchstabe, ein Name, ein Absatz, ein ganzes Buch). Dadurch dass Text eine beliebige Länge haben kann, die Reihenfolge der einzelnen Buchstaben jedoch ungeheuer wichtig ist (sonst wäre der Text ja nicht lesbar), spricht man von Zeichenketten. Die Übersetzung dieses Begriffs auf Englisch wäre character string. Deshalb findet sich in R für Text häufig die Bezeichnung character oder string die synonym verwendet werden.
3 Das Tidyverse
3.1 Vorbereitung
Pakete kann mensch bequem über die IDE installieren oder einfach mit einem R-Befehl:
Es ist wichtig, dass Sie für diesen Abschnitt alle Pakete installiert haben. – Aber keine Angst, sollten Sie ein Paket schon istalliert haben, dann wird es maximal auf die aktuelle Version upgedated!
3.2 Das tidyverse
tidyverse ist eine Sammlung von aufeinander abgestimmter Pakete für die Arbeit mit Daten.
Kernpakete:
Philosophie: Ein Datenzusammenhang pro Tabelle, eine Variable pro Spalte, eine Beobachtung pro Zeile; klare, “verb”-artige Funktionen.
Pipes: Verkette Schritte mit dem Base-Pipe |> (oder %>%) um lineare, lesbare Workflows zu bauen.
3.3 Anmerkung zu den Beispiel Daten “palmerpenguins”
Wir schauen uns die Daten der “Palmer Penguins” an.
Es wurden auf drei Inseln (“Biscoe”, “Dream” und “Torgersen”) der Antarktic drei Arten von Piguinen (“Adelie”, “Chinstrap” und “Gentoo”) beobachtet.1
3.4 Anmerkung zu den Beispiel Daten “palmerpenguins”
3.5 Beispiel
Wir haben schon ein wenig mit dem tidyverse gearbeitet, daher können Sie sich sicher denken, was wir hier ausgeben wollen:
3.6 Einlesen von Daten - Vorbereitung
Wir wollen nun Daten aus einer csv Datei einlesen. Dafür verwenden wir das Paket readr aus dem tidyverse. Es gibt aber auch Bord-Mittel. Schauen Sie sich gerne einmal die Befehle read.csv()" undread.csv2()` an.
Wir lesen nun die Daten “sales.csv” aus dem Verzeichnis “data” ein:
Und geben die Spaltennamen aus:
[1] "Region" "Country" "Item Type" "Sales Channel"
[5] "Order Priority" "Order Date" "Order ID" "Ship Date"
[9] "Units Sold" "Unit Price" "Unit Cost" "Total Revenue"
[13] "Total Cost" "Total Profit" Fragen an Sie: Sind diese Variabel-/Spaltennamen “schön”? - Was ist Ihre Meinung?
3.7 Einlesen von Daten und Variabelnamen vereinheitlich mit dem Paket janitor
Wie schauen die Spaltennamen nun aus?
3.8 Nochmal etwas Selektieren und Filtern
Wir erinnern uns: Mit select() selektieren wir Spalten. Mit filter() filtrieren wir Zeilen.
Erzeugen wir also eine neue Tabelle mit dem Namen df_small, die - nur die Spalten order_date, region und alle Spalten die mit total_ beginnen enthält. - Danach nur die Regionen “Europa” und “North America” enthält, die einen “total_revenue” von mit als 50000 Dollar haben:
3.9 Die Marge als neue Spalte margin hinzufügen
Fügen wir die Marge als neue Spalte margin hinzu. Die Marge ist der Quotient aus total_profit und total_revenue. Sortieren wir dann das Ergebnis absteigend (vgl: desc()) bzgl. der Spalte margin., speichern das Ergebnis in df_aug und geben anschließend die ersten 6 Zeilen der neuen Tabelle (head()) aus:
df_small |>
mutate(margin = total_profit / total_revenue) |>
arrange(desc(margin)) ->
df_aug
head(df_aug)# A tibble: 6 × 6
order_date region total_revenue total_cost total_profit margin
<chr> <chr> <dbl> <dbl> <dbl> <dbl>
1 4/23/2012 Europe 182825. 59960. 122865. 0.672
2 10/14/2014 Europe 856974. 281057. 575916. 0.672
3 2/2/2010 Europe 247956. 81321. 166635. 0.672
4 2/23/2015 Europe 1244708. 749701. 495008. 0.398
5 5/22/2017 Europe 793518 477944. 315574. 0.398
6 9/17/2012 Europe 3786589. 2280701. 1505888. 0.3983.10 Eine kurze Zusammenfassung der Daten
Wenn wir nun die mittleren Marge (mean_margin) und die Gesamtumsatz (total_rev) für jeweils unsere Regionen bestimmen möchten können wir den Befehll (summarize()) nutzen:
3.11 Eine andere Aufgabe
Etwas trickreicher müssen wir vorgehen, wenn wir für den Gesamtumsatz (total_revenue), die Gesamtkosten (total_costs) und den Gesamtgewinn (total_profit) die Durchschnitte und deren Standardabweichungen in eine neue Tabelle speichern wollen:
Wir beginnen mit einer Funktion, die uns für jeden übergebenen Wert eine Tabelle (tibble) mit dem Mittelwert mean und der Standardabweiung sd zurückliefert:
Diese Funktion nutzen wir und erzeugen eine neue Tabelle, in dem wir durch alle Spalten von total_revenue bis total_profit durchlaufen und diese der Funktion mean_sd() übergeben.
Das Ergebnis kommt dann in die Tabelle df_stats:
# A tibble: 1 × 6
total_revenue_mean total_revenue_sd total_cost_mean total_cost_sd
<dbl> <dbl> <dbl> <dbl>
1 1373488. 1460029. 931806. 1083938.
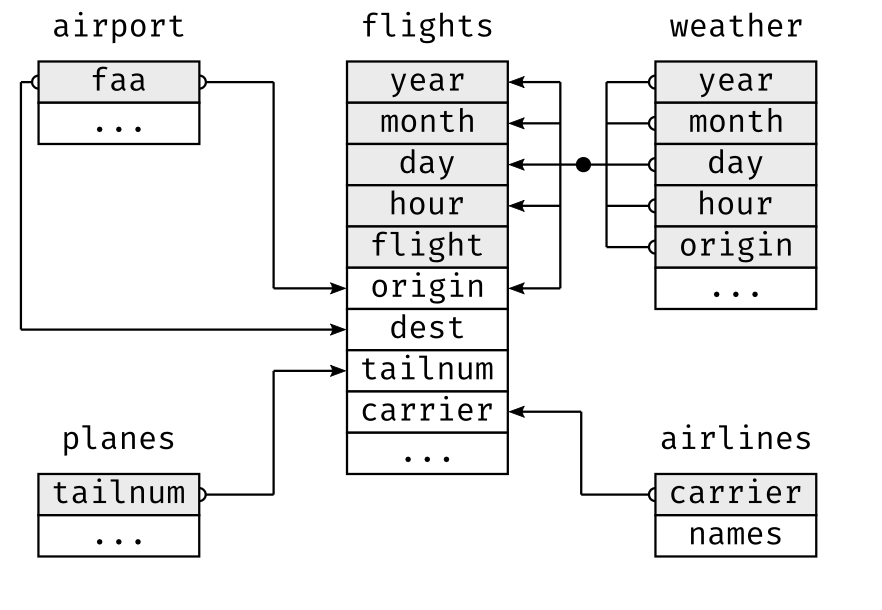
# ℹ 2 more variables: total_profit_mean <dbl>, total_profit_sd <dbl>3.12 Ein neues Beispiel: New York City Flüge aus dem Jahre 2013
3.13 Wir identifizieren Fluggesellschaft über ihre Abkürzungen
Dazu erstellen wir einen left join, so wie Sie ihn aus SQL kennen:
3.14 Nur die A320 Maschinen bitte!
Wir wollen wissen welche Fluggesellschaften im Februar mit einem Airbus A320 (egal welcher Baureihe) in New York City war:
3.15 Umformen mittels tidyr
Manchmal möchten wir Tabellen für Menschen lesbarer machen. Oder umgekehrt, für den Computer.
Hier kann das Paket tidyr helfen.
3.16 Gute Lesbarkeit für den Computer / Menschen
Stellen wir uns vor, wie möchten wissen wie viele Tiere welcher Art (species) auf welcher Insel (island) gesichtet wurden, dann geht das sehr gut mir dem Befehl count():
Das Problem ist, es ist nicht die Tabelle, die wir als Menschen gut lesen können. Daher nutzen wir die Funktion pivot_wider() aus dem Paket tidyr:
3.17 Visualisierung mit dem Paket ggplot2
3.17.1 Die Grundidee
Der erste Teil von “ggplot2” steht für “Grammar of Graphics”, also “Grammatik der Grafiken”.
Diese Idee Grafiken nicht einfach nur zu Programmieren, sondern den gesamten Prozess der Erstellung von Grafiken zu analysieren und für Nutzer:innen handhabbar zu machen geht u.a. auf das Buch “The Grammar of Graphics” von Leland Wilkinson zurück.
Es versucht die Bereiche
\text{Daten} + \text{Ästhetiken (aes)} + \text{Geome} + \text{Skalen} + \text{Facets} + \text{Themen}
zu kombinieren, so Grafiken einfach, klar zu beschreiben und den Computer daraus eine Graphik erstellen zu lassen.
Hinweis: Ein Geom ist, in Analogie zum Biom, ein eingeführter Begriff, der den “Normtyp” einer Graphik kennzeichnet.
3.18 Beispiel: Streudiagram Körpermasse vs. Flossenlänge:
3.19 Beispiel: Flossenlänge vs. Schnabellänge
3.20 Beispiel: Histogramm der Körpermasse:
3.21 Beispiel: Boxplot der Körpermasse nach Art
3.22 Datum mit dem Paket lubridate
Die R-Bibliothek lubridate wird verwendet, um Datums- und Zeitangaben einfach zu verarbeiten und zu manipulieren.
Wir nutzen hier die Befehle make_datetime() um aus den Datumsangaben eine datetime() zu bilden.
Und den Befehl wday(), der und aus einem Datum den Wochentag berechnet.
3.23 Vorbereitung
Wir schauen uns jetzt nur die Flüge im Januar an und fügen den Wochentag (wday) und die Abflugszeit (dep_datetime) hinzu:
3.24 Säulendiagramm: Flüge nach Wochentag im Januar
3.25 Streudiagramm Ankunfts- vs. Abflugverzögerungen im Januar
3.26 Aufgaben
Erstellen Sie eine Tabelle für den August mit dem Namen aug_flights
Fügen Sie von den Fluggesellschaftskürzeln auch die entsprechenden Namen der Fluggesellschaften in die Tabelle aug_flights ein.
Geben Sie die Anzahl der Flüge im August nach Wochentagen sortiert als Säulendiagramm aus.
4 Datenbanken als Datenquelle
4.1 Die Verbindung zu einer Datenbank
Für die Verbindung zu einer Datenbank ist das Paket DBI zuständig. Sie brauchen zusätzlich einen Treiber. Dieser kann ein ODBC-Treiber sein.
ODBC (englisch für etwa Offene Datenbank-Verbindungsfähigkeit) ist, so sagt Wikipedia, “eine standardisierte Datenbankschnittstelle, die SQL als Datenbanksprache verwendet. Es bietet also eine Programmierschnittstelle (API), die es einem Programmierer erlaubt, seine Anwendung relativ unabhängig vom verwendeten Datenbankmanagementsystem (DBMS) zu entwickeln, wenn dafür ein ODBC-Treiber existiert.”
Ebenso sind aber auch direkte R-Treiben möglich, wie etwa RPostgreSQL, RSQLite und RMariaDB, die nur bestimmte Datenbanken unterstützen. (RMariaDB z.B. neben MariaDB auch MySQL)
4.2 Für den Einstieg eine SQLite Datenbank
Um mit einer Datenbank zu Arbeiten gibt es mehrere Wege. Wir wollen mit einer SQLite Datenbank beginnen.
Dazu brauchen ein paar Pakete:
Wir können direkt mit SQLite arbeiten oder aber via einer ODBC-API. Für letzteres müssen wir ein paar Pakete in den Sopeicher laden:
Danach erstellen wir die eigentliche Verbindung zur Datenbank mit dem Befehl dbConnect():
Die Angabe von “Database=sqlite-db/mitarbeiter.db” referenziert auf die Datei “mitarbeiter.db” im Verzeichnis “sqlit-db” in der eine solche Datenbank liegt.
4.3 Alternativ eine Verbindung direkt mit dem RSQLite-Treiber
Wir können mit dem Paket RSQLite direkt eine Verbindung mit der Datenbank herstellen,. Nachdem wir die notwendigen Pakete geladen haben:
Erzeugen wir nun die Verbindung zur Datenbank mit Hilfe des dbConnect-Befehls:
Wieder wird hier die Datei im Verzeichnis “sqlite-db” angegeben.
4.4 Auslesen aus der Datenbank
Aus der Datenbank können wir komplette Tabellen herunterladen und lokal speichern:
# Tabelle einlesen
dbReadTable(con, "mitarbeiter") |> # Laden der Tablle 'mitarbeit'
as_tibble() -> # Umwandeln in de tibbel-Datentabelle
daten_tib # Speichern der Tabele in der Variable daten_tib
# Verbindung schließen
dbDisconnect(con)
# Informationen über die Tabelle ausgeben:
glimpse(daten_tib)Rows: 20
Columns: 7
$ id <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, …
$ title <chr> "Dipl.-Ing.", "Meike", "", "", "", "Stanislav", "Univ.P…
$ vorname <chr> "Karina", "van", "Siegried", "Stanislaus", "Hermann-Jos…
$ nachname <chr> "Löffler", "der", "Baum", "Tschentscher-Heß", "Kühnert"…
$ abteilung <chr> "IT", "Finanzen", "Vertrieb", "Marketing", "IT", "Marke…
$ eintrittsdatum <chr> "2011-11-04", "2020-08-25", "2019-04-07", "2011-07-10",…
$ gehalt <dbl> 48836.14, 44175.54, 82034.03, 52670.45, 73735.47, 55386…4.5 Weiter in R
Mit dieser Tabelle können wir wie gewohnt arbeiten in R arbeiten, denn wir haben die lokal gespeichert:
# Daten in data.table anzeigen
# Optional: Daten filtern und transformieren mit tidyverse/dplyr
daten_tib |>
filter(abteilung == "HR") |> # Nur die Abteilung HR
as_tibble() -> # In ein Tibble umwandeln
daten_tib_gefiltert # und lokal speichern
# Informationen über die Tabelle ausgeben:
glimpse(daten_tib_gefiltert)Rows: 3
Columns: 7
$ id <int> 14, 18, 20
$ title <chr> "", "", ""
$ vorname <chr> "Rolf-Peter", "Irmela", "Harro"
$ nachname <chr> "Beyer", "Striebitz", "Girschner"
$ abteilung <chr> "HR", "HR", "HR"
$ eintrittsdatum <chr> "2019-07-13", "2016-09-25", "2018-09-22"
$ gehalt <dbl> 35483.28, 41833.14, 44093.324.6 Auslesen in der Datenbank
Alternativ können wir auch eine Verbindung mit der Tabelle in der Datenbank herstellen, ohne immer gleich alle Daten lokal herunter zu laden! Mit dem Befehl tbl()erstellen wir quasi eine Verlinkung her.
# Verbindung zur SQLite-Datenbank herstellen
con <- dbConnect(SQLite(), "sqlite-db/mitarbeiter.db")
# Link auf die Tabelle in der Datenbank
daten_db <-tbl(con, "mitarbeiter")
# Informationen über die Tabelle ausgeben:
glimpse(daten_db)Rows: ??
Columns: 7
Database: sqlite 3.50.4 [/Users/norman/Documents/GitHub/FOM-NM-Lehre/ProgrammierenInR-neu/Workshop/sqlite-db/mitarbeiter.db]
$ id <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, …
$ title <chr> "Dipl.-Ing.", "Meike", "", "", "", "Stanislav", "Univ.P…
$ vorname <chr> "Karina", "van", "Siegried", "Stanislaus", "Hermann-Jos…
$ nachname <chr> "Löffler", "der", "Baum", "Tschentscher-Heß", "Kühnert"…
$ abteilung <chr> "IT", "Finanzen", "Vertrieb", "Marketing", "IT", "Marke…
$ eintrittsdatum <chr> "2011-11-04", "2020-08-25", "2019-04-07", "2011-07-10",…
$ gehalt <dbl> 48836.14, 44175.54, 82034.03, 52670.45, 73735.47, 55386…4.7 Auslesen in der Datenbank
# Optional: Daten filtern und transformieren mit tidyverse/dplyr
daten_db |>
filter(abteilung == "HR") |>
as_tibble() -> daten_db_gefiltert
glimpse(daten_db_gefiltert)Rows: 3
Columns: 7
$ id <int> 14, 18, 20
$ title <chr> "", "", ""
$ vorname <chr> "Rolf-Peter", "Irmela", "Harro"
$ nachname <chr> "Beyer", "Striebitz", "Girschner"
$ abteilung <chr> "HR", "HR", "HR"
$ eintrittsdatum <chr> "2019-07-13", "2016-09-25", "2018-09-22"
$ gehalt <dbl> 35483.28, 41833.14, 44093.324.8 Direktes Arbeiten mit der Datenbank
Es gibt einige Befehle, mit denen wirk dirket mit der Datenbank kommunizieren können:
Zum Beispiel können wir die Liste aller Tabellen in der Datenbank abfragen:
4.9 SQL oder d(b)plyr ?
Das Paket dplyr merkt, wenn es auf Datenbank-Tabellen arbeitet und übersitzt, mit Hilfe des Paketes dbplyr im Hintergrund die R-Befehle in SQL-Befehl! Schauen wir uns das einmal am Beispiel des filter()-Befehls an:
<SQL>
SELECT `mitarbeiter`.*
FROM `mitarbeiter`
WHERE (`abteilung` = 'HR')Sie sehen, die R-Befehl wird hier in eine SQL-Anweisung übersetzt. Diese wird an die Datenbank gesendet und die Ergebnisse wieder nach R transformiert.
4.10 LIVE!
… Weiter in der Live-Demo …
5 Quarto - Für Berichte
5.1 Einleitung oder die Anwort auf: Was ist Quarto?
Quarto ist eine mehrsprachige, modernisierte Version von R Markdown und umfasst Dutzende neuer Funktionen und Möglichkeiten, während es gleichzeitig die meisten vorhandenen Rmd-Dateien (also R markdown Dateien) ohne Modifikation übersetzen kann.
In diesem Workshop zeigen ich Ihnen, wie Sie Quarto verwenden. Sie werden Code und Markdown bearbeiten. Sie lernen, wie Sie mit jedem Rechendokument (z.B. R oder Python) umgehen, und das gerenderte Dokument im Viewer-Tab während Ihrer Arbeit anzeigen.
5.2 Was ist Quarto nicht?
Quarto ist kein Wunderwerk.
Es vereinfacht u.U. einiges in ihrem Arbeitsfluss. Aber um das zu erreichen nimmt es Ihnen auch Entscheidung z.B. über das Layout ab. – Vieles können Sie aber steuern und ggf. (mit Mühe) Ihren Bedürfnissen anpassen.
Wer Freiheit will muss also immer noch Extraarbeit in Projekte investieren.
Wer aber mit vorgefertigten Dokumentenfassungen zu frieden ist, hat mit Quarto ein Veröffentlichungssystem gefunden, das viele Abreitsschritte vereinfacht.
Quarto ist auch kein Selbstläufer.
Weder für Dozent:innen, noch für Lernende! Beide brauchen Anleitung und Hilfestellung! Ohne Schulung können wir hier keine guten Ergebnisse erwarten.
5.3 Die Installation von Quarto
Auf der Seite Get Started Seite von Quarto können Sie die Quarto CLI für Ihr Betriebssystem herunterladen und Installieren.
Zum Umwandeln von Quarto Dokumenten in R Studio bzw. posit.cloud brauchen Sie noch das R Paket
quarto, welches Sie wie folgt in R installieren können:
- Falls Sie LaTeX benutzen wollen, können Sie es aus dem Terminal wie folgt installieren:
- Im Verzeichnis
setuphabe ich ihnen mitsetup.Rein kleines R Skript erstellt, welches die notwendigsten Pakete installiert.
5.4 Der Weg der Quarto markdown Datei zum Ausgabeformat
Wir sehen hier die Abhöngigkeit von Quarto (eben so wie wir es von R markdown kennen) von pandoc!
Auch wenn pandoc sich selber vorstellt mit
If you need to convert files from one markup format into another, pandoc is your swiss-army knife.
wissen wir , dass auch ein Schweizer Taschenmesser nicht alle Probleme optimal lösen kann.
Um es klar zu fomulieren:
Wer nur ein Dokument erstellen will, mit einem festen Inhalt und dabei keine Berechnung (in R oder Python) braucht ist fast immer mit anderen Werkzeugen besser bedient.
5.5 Der Weg der Quarto markdown Datei zum Ausgabeformat (Fortsetzung)
Wer aber aus einem Dokument u.U. mehrere Zielformate bespielen will. Wer umfangreiche Berechungen (in R oder Python) hat die dynamische Änderungen nach sich ziehen. Wer einen Weg sucht dieses, auf vorgefertigten Wegen, ohne viel mehr zu lernen erreichen will, der kommt am Quarto kaum vorbei.
Aber: TANSTAAFL2
Da Quarto auf pandoc basiert hängen wir sehr stark von den Fähigkeit von pandoc ab. Das zeigt sich unter anderem bei den Ausgabeformaten PDF und Beamer. Diese benutzen neben pandoc eben auch eine funktionierende (und gewartete) TeX (und LaTeX) Version!
In der R-Welt können wir das u.U. mit dem Paketen tinytex erhalten. Alternative können wir auch TeX (und damit auch LaTeX) über eine direkte Installation nutzbar machen. Informationen zu einer solchen Installation erhalten Sie bei Dante e.V..
5.6 Die Ausgabeformate (ein Überblick)
5.7 Dokumente
- HTML
- PDF
- MS Word
- OpenOffice
- ePub
5.8 Presentations
- Revealjs
- PowerPoint
- Beamer
5.9 Markdown
- GitHub (GitHub Flavored Markdown (GFM))
- CommonMark
- Hugo
- Docusaurus
- Markua
5.10 Wikis
- MediaWiki
- DokuWiki
- ZimWiki
- Jira Wiki
- XWiki
Und noch eine ganze Reihe weitere Formate. (Siehe hier)
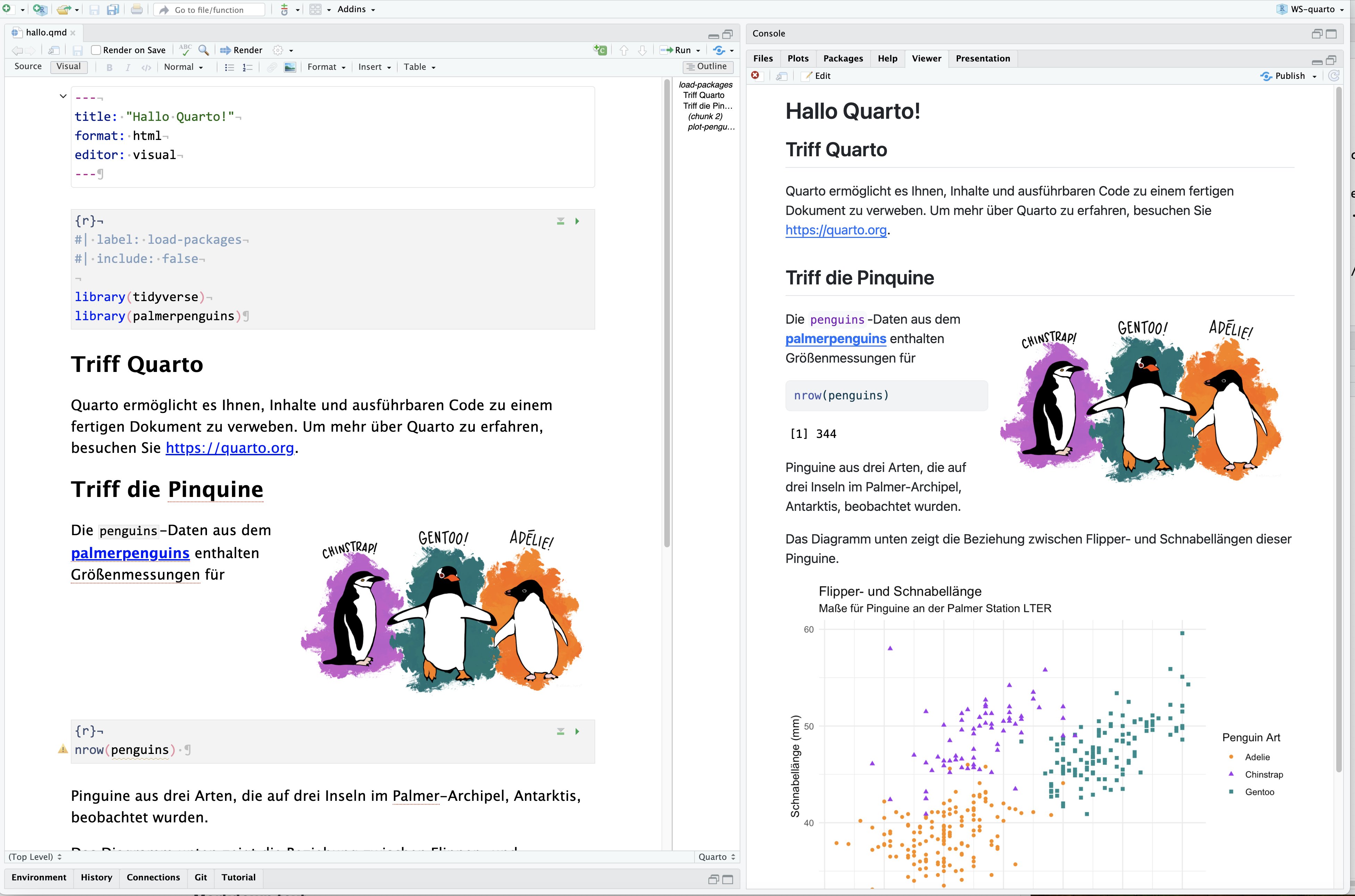
5.11 Ein erstes Dokument: Beispiel 1 - hallo.qmd
Der folgende Text ist ein Quarto-Dokument mit der Erweiterung .qmd (links) sowie die gerenderte Version als HTML (rechts). Sie können es auch in andere Formate wie PDF, MS Word usw. rendern.
5.12 Beispiel 1 (Fortsetzung)
Dies ist das Grundidee für die Veröffentlichung mit Quarto – nehmen Sie ein Quelldokument und wandeln3 Sie es in eine Vielzahl von Ausgabeformaten mit Quarto um.
5.13 Umwandeln oder rendern
Die Schaltfläche “Rendern” in der RStudio-IDE (oder posit.cloud) können Sie nutzen um die Datei zu rendern und die Ausgabe mit einem einzigen Klick oder Tastaturkürzel (⇧⌘K) zu betrachten.
Sie finden Sie Schaltfläche oben im Texteditor:
Sie können auch beim Speichern einer Datei diese automatisch rendern lassen. Dazu können Sie die Option “Rendern beim Speichern” in der Symbolleiste des Editors aktivieren. Die Vorschau wird aktualisiert, sobald Sie das Dokument erneut rendern.
Die nebeneinander angeordnete Vorschau funktioniert sowohl für HTML- als auch PDF-Ausgaben.
5.14 Umwandeln oder rendern (Fortsetzung)
Umwandeln mittels R-Konsole und dem R-Paket quarto der Datei in ein PDF-Dokument:
Umwandeln aus dem Terminal mittels der Quarto CLI:
Beim Rendern erzeugt Quarto eine neue Datei, die den ausgewählten Text, Code und deren Ergebnisse aus der .qmd-Datei enthält.
Die neue Datei kann ein HTML-, PDF-, MS Word-Dokument, eine Präsentation, eine Website, ein Buch, ein interaktives Dokument oder ein anderes Format sein.
5.15 Die Quarto CLI
Das Quarto CLI4 ermöglicht es auch außerhalb einer IDE5 bzw. R mit einem Quarto Markdown Dokument zu arbeiten.
Hilfe erhalten Sie mit:
Ein (ganzes) Quarto Projekt können Sie wie folgt erstellen:
Mit diesem Befehl wird ein neues Verzeichnis mit dem Namen “Neues_Projekt” erzeugt.
5.16 Die Quarto CLI (Fortsetzung)
In dem Verzeichnis “Neues_Projekt” sind nun die folgenden Dateien enthalten:
“Neues_Projekt.Rproj” (eine Projekt Datei für RStudio/ posit.cloud),
“Neues_Projekt.qmd” (ihre Startdatei für das eigene Dokument) und
“_quarto.yml” (ein YAML-Datei für alle qmd Dateien im Verzeichnis, dazu später mehr)
5.17 Die Quarto CLI (Fortsetzung)
Eine Quarto Markdown Datei (typischerweise mit der Endung .qmd) kann mittels Quarto CLI umgewandelt werden:
Sie können für die Ausgabe auch ein Ausgabeformat angeben (hier z.B. ´html´):
Wenn Sie anstatt eines Dokumentes ein Quarto Verzeichnis angeben, wir das gesamte Projekt übersetzt:
5.18 Der (optionale) YAML-Kopf
Ein (optionaler) YAML6-Kopf wird von drei Bindestrichen (- - -) an beiden Enden begrenzt.
Beim Übersetzen wird der Titel “Hallo Quarto!” oben im übersetzen Dokument mit einer größeren Schriftgröße als der Rest des Dokuments angezeigt. Die anderen beiden YAML-Felder geben an, dass die Ausgabe im HTML-Format erfolgen soll und das Dokument standardmäßig im visuellen Editor geöffnet werden soll.
5.19 Der (optionale) YAML-Kopf (Fortsetung)
Die grundlegende Syntax von YAML verwendet Schlüssel-Wert-Paare im Format “Schlüssel: Wert” (engl. key: value).
YAML-Felder, die häufig in Kopfzeilen von Dokumenten gefunden werden, umfassen Metadaten wie Autor (author), Untertitel (subtitle), Datum (date) sowie Anpassungsoptionen wie Thema (theme), Schriftfarbe (fontcolor), Abbildungsweite (fig-width) usw.
---
title: "Hallo Quarto"
subtitle: "Eine kurze Einführung"
date: 2024-06-06
author: "Norman Markgraf"
format:
html: default
docx: default
---
Alle verfügbaren YAML-Felder für HTML-Dokumente finden Sie hier.
5.20 Der (optionale) YAML-Kopf (Fortsetung)
Die verfügbaren YAML-Felder variieren je nach Zielformat. Je nach Zielformat finden Sie hier den Link zu den wichtigsten YAML-Feldern:
- PDF-Dokumente,
- MS Word und
- OpenDocument Format (für LibreOffice oder Apache OpenOffice).
5.21 Markdown Basics
Quarto basiert auf pandoc und verwendet dessen Variante von Markdown als zugrunde liegende Dokumentensyntax. Pandoc-Markdown ist eine erweiterte und leicht überarbeitete Version der Markdown-Syntax von John Gruber.
A Markdown-formatted document should be publishable as-is, as plain text, without looking like it’s been marked up with tags or formatting instructions. – John Gruber
Im folgenden finden Sie Beispiele für die am häufigsten verwendete Markdown-Syntax.
Eine vollständige Dokumentation von Pandocs Markdown finden Sie hier.
5.22 Text Formatierung
| Markdown Syntax | Ausgabe |
|---|---|
| *kursiv*, _kursiv_, **fett**, ***fett kursiv*** | kursiv, kursiv, fett, fett kursiv |
| superscript^2^ / subscript~2~ | superscript2 / subscript2 |
| ~~durchgestrichen~~ | |
| `verbatim code` | verbatim code |
5.23 Kopfzeilen
| Markdown Syntax | Ausgabe |
|---|---|
| # Kopf 1 | Kopf 1 |
| ## Kopf 2 | Kopf 2 |
| ### Kopf 3 | Kopf 3 |
| #### Kopf 4 | Kopf 4 |
| ##### Kopf 5 | Kopf 5 |
5.24 Links & Bilder
| Markdown Syntax | Ausgabe |
|---|---|
<http://quarto.org> |
http://quarto.org |
[Quarto](http://quarto.org) |
Quarto |
 |
 |
[](https://quarto.org) |
 |
5.25 Listen I/II
5.27.2
- ungeortnete Liste
- Unterpunkt 1
- Unterpunkt 2
- Unter-Unterpunkt 1
5.27.4
Punkt 2
Weiter in diesem Punkt mit 4 Leerzeichen am Zeilenanfang.
5.28 Listen II/II
5.30.2
- Eine Liste die hier starte
geht weiter
- nach einer Unterbrechung
5.30.4
- Eine Liste
- Gefolgt von einer zweiten Liste
5.31 Listen (Wichtig!)
Im Gegensatz zu anderen Markdown-Renderern (insbesondere Jupyter und GitHub) müssen Listen in Quarto eine vollständige Leerzeile oberhalb und unterhalb der Liste haben!
Andernfalls wird die Liste nicht als Liste erkannt und nicht in Listenform gerendert.
Sie erscheint damit als normaler Text in einer einzigen Zeile.
5.32 Tabellen
5.33 Quellcode
Sie können mit ``` einen Codeblock begrenzen:
Durch die Angabe einer Sprache aktivieren Sie die passende Syntax-Herforhebung:
Pandoc unterstützt die Syntaxhervorhebung für über 140 verschiedene Programmiersprachen. Wenn Ihre Sprache nicht unterstützt wird, können Sie die Standardsprache default verwenden, um eine ähnliche visuelle Darstellung zu erhalten:
5.34 Roh-Inhalte
Ein Roh-Inhalt kann direkt eingefügt werden ohne dass Quarto ihn übersetzt. Dazu wird ein pandoc raw-Attribut verwendet.
Ein raw-Block beginnt mit ```{= gefolgt von einem Format und schließt mit }, z. B. hier ist ein raw-HTML-Block:
Oder hier für raw-(La)TeX Blöcke:
Sogenannter inline-Code wird wie folgt einfügt:
5.35 Mathematische Formeln
Verwenden Sie $-Begrenzer für Inline-Formel und $$-Begrenzer für Displaystyle-Formeln. Zum Beispiel:
| Markdown Syntax | Ausgabe |
|---|---|
| Inline math: $E = mc^{2}$ | Inline math: E = mc^{2} |
| Display math: $$E = mc^{2}$$ | Display math: E = mc^{2} |
Für eigene TeX-Macros können Sie den $$-Begrenzer in einem .hidden Block nutzen:
5.36 Mathematische Formeln
Beachten Sie:
Für HTML benutzt pandoc in der Regel MathJax. Ich empfehle Ihnen aber KaTeX zu nutzen. Dazu können Sie im YAML-Kopf die folgende Zeile einfügen:
5.37 Diagramme
Quarto unterstützt das Einbetten von Mermaid- und Graphviz-Diagrammen nativ. Dies ermöglicht es Ihnen, Flussdiagramme, Sequenzdiagramme, Zustandsdiagramme, Gantt-Diagramme und mehr mit einer Klartextsyntax zu erstellen, die von Markdown inspiriert ist.
Zum Beispiel betten wir hier ein mit Mermaid erstelltes Flussdiagramm ein:
Mehr finden sie hier.
5.38 Videos
Videos können in Dokumente einfügen, indem Sie den {{< video >}}-Shortcode verwenden. Als Beispiel betten wir hier ein YouTube-Video ein:
Videos können auf Videodateien (z.B. MPEG) verweisen oder Links zu auf YouTube, Vimeo oder Brightcove veröffentlichten Videos sein.
Mehr zu dem Thema finden sie hier.
5.39 Seitenumbrüche
Der pagebreak-Shortcode ermöglicht es Ihnen, einen nativen Seitenumbruch in ein Dokument einzufügen (z.B. in LaTeX wäre dies ein \newpage, in MS Word ein docx-nativer Seitenumbruch, in HTML eine page-break-after: always CSS-Direktive, usw.):
Native Seitenumbrüche werden für HTML, LaTeX, Context, MS Word, Open Document und ePub unterstützt (für andere Formate wird ein Form-Feed-Zeichen \f eingefügt).
5.40 Callout Blöcke
Mehr zu diesem Thema Callout Blocks finden sie hier.
5.41 Die Datei “_quarto.yml”
Die Datei “_quarto.yml” im Hauptverzeichnis kann zusätzliche Optionen im YAML Syntax enthalten und wird vor dem jeweiligen YAML-Kopf der qmd-Datei geladen und verarbeitet.
Die in der Datei hinterlegten Optionen werden somit von jeder in dem Verzeichnis gelegenden qmd-Datei übernommen.
Ein Beispiel einer “_quarto.yaml” Datei:
title: |
| Eine kurze Einführung
| Workshop: Quarto
institute: "FOM"
author: "Dipl.-Math. Norman Markgraf"
lang: de
format:
html:
html-math-method: katex
So können Sie aus einem Verzeichnis unterschiedliche Dokument, zum Beispiel für Tagesfolien einer Veranstaltungsreihe, immer mit den selben YAML Informationen füttern.
5.42 Beispiel 2: Mehrere qmd Dateien mit einer “_quarto.yaml” Datei
Im Verzeichnis “Beispiel 2” haben Sie eine kleine Anwendung für eine “_quarto.yaml” Datei.
In dem Ordner befinden sich zwei ‘Vorlesungs’-Dateien. Beide gehören zur selben Vorlesung und haben viele der Meta-Daten (u.a. Titel, Sub-Titel und Autor) gemeinsam. Daher ist es sinnvoll diese aus den qmd-Dateien in die “_quarto.yml” auszulagern.
Übung:
Gehen Sie via Terminal in das Verzeichnis “Beispiel 2” und nutzen Sie die Quarto CLI um alle Vorlesungsdateien zu rendern.
Tipp:
Sie benötigen zum Rendern nur einen Befehl!
6 ShinyApp - Für Dashboards
6.1 Was ist eine Shiny-App?
Eine Shiny-App ist eine interaktive Webanwendung, die mit der Programmiersprache R erstellt wird. Das Shiny-Paket ermöglicht es, benutzerfreundliche Web-Apps zu bauen, die R-Code im Hintergrund ausführen und dabei Daten, Visualisierungen oder Modelle dynamisch an die Benutzereingaben anpassen.
6.2 Kernpunkte zu Shiny-Apps:
Sie bestehen aus zwei Hauptkomponenten:
User Interface (UI): Definiert das Aussehen und die Bedienungselemente für den Benutzer.
Server: Enthält die Logik, wie auf Benutzereingaben reagiert und Ausgaben generiert werden.
6.3 Mehr
Shiny nutzt reaktives Programmieren, sodass Änderungen an den Eingaben automatisch die entsprechenden Ausgaben aktualisieren.
Shiny-Apps laufen lokal im Browser oder online auf einem Shiny-Server und benötigen keine Kenntnisse in HTML, CSS oder JavaScript, da dies vom Paket automatisch gehandhabt wird.
Damit lassen sich z.B. interaktive Dashboards, Visualisierungen, Berichte oder datengetriebene Webtools schnell und unkompliziert umsetzen.
Kurz gesagt: Eine Shiny-App verbindet die R-Programmierung mit modernen interaktiven Webtechnologien, um leicht bedienbare und dynamische Anwendungen für Datenanalyse und Visualisierung bereitzustellen.
6.4 Ein Beispiel
Live-Beispiel im Verzeichnis “KleineShinyApp” ..
Wir sind am Ende …
… unserer kleinen Reise der “Sechs Dinge über R” angekommen.
Sicher sind viele Fragen offen. – Falls Sie noch nicht gleich eine zur Hand haben, aber später dennoch Fragen wollen:
Sie erreichen mich unter norman.markgraf@fom-net.de!
Die (jeweils) aktuelle Version dieses Workshops finden Sie im GutHub-Repository unter https://github.com/FOM-NM-Lehre/ProgrammierenInR.git
WICHTIG!
Copyright in 2025 by Norman Markgraf all right reserved!
Weitergabe im ganzen erwünscht! Verwendung, auch auszugsweise, unterliegt der nebenstehenden Lizenzvereinbarung!
Vielen Dank für Ihre Aufmerksamkeit.
Just for the records
─ Session info ───────────────────────────────────────────────────────────────
setting value
version R version 4.5.1 (2025-06-13)
os macOS Sequoia 15.6.1
system x86_64, darwin20
ui X11
language (EN)
collate en_US.UTF-8
ctype en_US.UTF-8
tz Europe/Berlin
date 2025-09-08
pandoc 3.7.0.2 @ /usr/local/bin/ (via rmarkdown)
quarto 1.7.34 @ /usr/local/bin/quarto
─ Packages ───────────────────────────────────────────────────────────────────
package * version date (UTC) lib source
bit 4.6.0 2025-03-06 [1] CRAN (R 4.5.0)
bit64 4.6.0-1 2025-01-16 [1] CRAN (R 4.5.0)
blob 1.2.4 2023-03-17 [1] CRAN (R 4.5.0)
cachem 1.1.0 2024-05-16 [1] CRAN (R 4.5.0)
cellranger 1.1.0 2016-07-27 [1] CRAN (R 4.5.0)
cli 3.6.5 2025-04-23 [1] CRAN (R 4.5.0)
crayon 1.5.3 2024-06-20 [1] CRAN (R 4.5.0)
DBI * 1.2.3 2024-06-02 [1] CRAN (R 4.5.0)
dbplyr 2.5.0 2024-03-19 [1] CRAN (R 4.5.0)
digest 0.6.37 2024-08-19 [1] CRAN (R 4.5.0)
dplyr * 1.1.4 2023-11-17 [1] CRAN (R 4.5.0)
evaluate 1.0.5 2025-08-27 [1] CRAN (R 4.5.1)
farver 2.1.2 2024-05-13 [1] CRAN (R 4.5.0)
fastmap 1.2.0 2024-05-15 [1] CRAN (R 4.5.0)
forcats * 1.0.0 2023-01-29 [1] CRAN (R 4.5.0)
generics 0.1.4 2025-05-09 [1] CRAN (R 4.5.0)
ggplot2 * 3.5.2 2025-04-09 [1] CRAN (R 4.5.0)
glue 1.8.0 2024-09-30 [1] CRAN (R 4.5.0)
gtable 0.3.6 2024-10-25 [1] CRAN (R 4.5.0)
here * 1.0.1 2020-12-13 [1] CRAN (R 4.5.0)
hms 1.1.3 2023-03-21 [1] CRAN (R 4.5.0)
htmltools 0.5.8.1 2024-04-04 [1] CRAN (R 4.5.0)
janitor * 2.2.1 2024-12-22 [1] CRAN (R 4.5.0)
jsonlite 2.0.0 2025-03-27 [1] CRAN (R 4.5.0)
knitr 1.50 2025-03-16 [1] CRAN (R 4.5.0)
lifecycle 1.0.4 2023-11-07 [1] CRAN (R 4.5.0)
lubridate * 1.9.4 2024-12-08 [1] CRAN (R 4.5.0)
magrittr 2.0.3 2022-03-30 [1] CRAN (R 4.5.0)
memoise 2.0.1 2021-11-26 [1] CRAN (R 4.5.0)
odbc * 1.6.2 2025-08-28 [1] CRAN (R 4.5.1)
pillar 1.11.0 2025-07-04 [1] CRAN (R 4.5.1)
pkgconfig 2.0.3 2019-09-22 [1] CRAN (R 4.5.0)
purrr * 1.1.0 2025-07-10 [1] CRAN (R 4.5.1)
R6 2.6.1 2025-02-15 [1] CRAN (R 4.5.0)
RColorBrewer 1.1-3 2022-04-03 [1] CRAN (R 4.5.0)
Rcpp 1.1.0 2025-07-02 [1] CRAN (R 4.5.1)
readr * 2.1.5 2024-01-10 [1] CRAN (R 4.5.0)
readxl * 1.4.5 2025-03-07 [1] CRAN (R 4.5.0)
rlang 1.1.6 2025-04-11 [1] CRAN (R 4.5.0)
rmarkdown 2.29 2024-11-04 [1] CRAN (R 4.5.0)
rprojroot 2.1.1 2025-08-26 [1] CRAN (R 4.5.1)
RSQLite * 2.4.3 2025-08-20 [1] CRAN (R 4.5.1)
rstudioapi 0.17.1 2024-10-22 [1] CRAN (R 4.5.0)
scales 1.4.0 2025-04-24 [1] CRAN (R 4.5.0)
sessioninfo 1.2.3 2025-02-05 [1] CRAN (R 4.5.0)
snakecase 0.11.1 2023-08-27 [1] CRAN (R 4.5.0)
stringi 1.8.7 2025-03-27 [1] CRAN (R 4.5.0)
stringr * 1.5.1 2023-11-14 [1] CRAN (R 4.5.0)
tibble * 3.3.0 2025-06-08 [1] CRAN (R 4.5.0)
tidyr * 1.3.1 2024-01-24 [1] CRAN (R 4.5.0)
tidyselect 1.2.1 2024-03-11 [1] CRAN (R 4.5.0)
tidyverse * 2.0.0 2023-02-22 [1] CRAN (R 4.5.0)
timechange 0.3.0 2024-01-18 [1] CRAN (R 4.5.0)
tzdb 0.5.0 2025-03-15 [1] CRAN (R 4.5.0)
utf8 1.2.6 2025-06-08 [1] CRAN (R 4.5.0)
vctrs 0.6.5 2023-12-01 [1] CRAN (R 4.5.0)
vroom 1.6.5 2023-12-05 [1] CRAN (R 4.5.0)
withr 3.0.2 2024-10-28 [1] CRAN (R 4.5.0)
xfun 0.53 2025-08-19 [1] CRAN (R 4.5.1)
yaml 2.3.10 2024-07-26 [1] CRAN (R 4.5.0)
[1] /Library/Frameworks/R.framework/Versions/4.5-x86_64/Resources/library
* ── Packages attached to the search path.
──────────────────────────────────────────────────────────────────────────────Fußnoten
Paper zu den Daten finden Sie hier
Nach Robert A. Heinlein aus seinem Roman
Revolte auf Luna(1966) : „there ain’t no such thing as a free lunch”Dieses
umwandelnnennen wir auchrendern. ;-)Quarto CLI steht für Quarto Command Line Interface, die Kommandozeileschnittstelle.
IDE steht für Integrated Development Environment, Integrierte Entwicklungsumgebung
YAML steht für “YAML Ain’t Markup Language” und dient hier dazu den Übersetzungs- bzw. Bearbeitungsprozess durch zusätzlichen Optionen zu steuern. Vgl. auch YAML bei Wikipedia
Workshop: Sechs Dinge über R | Dipl.-Math. Norman Markgraf